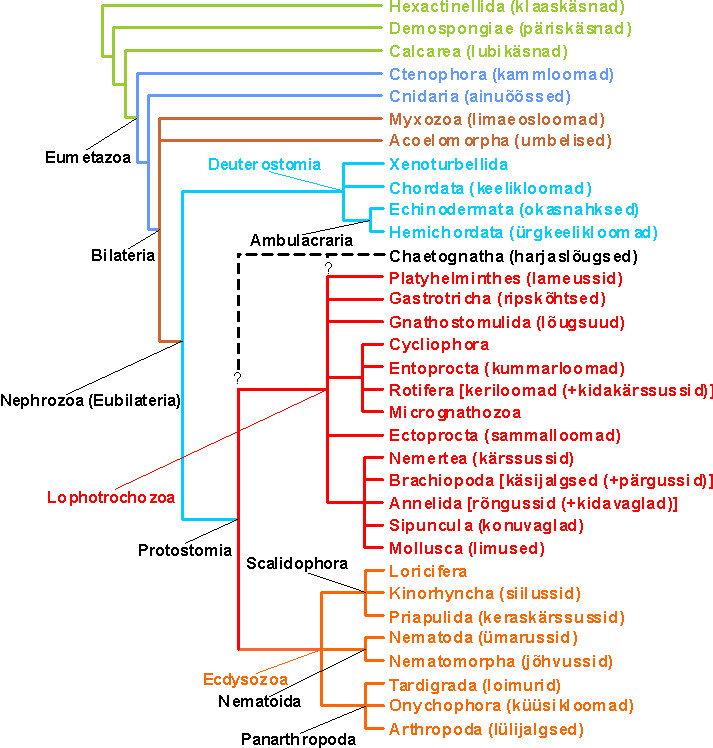
NB! Kohati vananenud arusaam loomade fülogeneesist, vajab hädasti uuendamist.
Joonis 1. Hulkraksete loomade fülogenees. Minu arvamuse kohaselt võiks ta selline välja näha. Just sellist fülogeneesi kinnitavaid andmeid on vähe ja eks aeg näitab kui palju sellest ka tõeks osutub. Kladogrammilt on välja jäetud Placozoa, Orthonectida ja Rhombozoa. Täpsemalt nendest ja teistest taksonitest allpool.
Suurelt osalt sarnaneb ülaltoodud fülogenees Halanych (2004) nägemusega. Giribet (2002) on ülaltoodud Lophotrochozoa (sensu Halanych, 2004) klaadi nimetuse asemel kasutanud nime Spiralia ning Lophotrochozoa reserveerinud väiksemale hulgale hõimkondadele. Kuna termin Lophotrochozoa on defineeritud fülogeneetiliselt, siis on seda üks-üheselt võimalik piiritleda teades piisavalt definitsiooni sisse arvatud taksonite ja nende lähisugulaste vahelisi fülogeneetilisi suhteid. Antud juhul on definitsioon järgmine: Lophotrochozoa on klaad, kuhu kuuluvad hõimkondade Brachiopoda (+Phoronida) ja Ectoprocta (moodustavad kunstliku rühma Lophophorata) ning Mollusca ja Annelida (kuuluvad Trochozoa hulka) viimane ühine eellane ning nende kõik järeltulijad (Halanych et al., 1995). Paistab, et Lophotrochozoa koostis oleneb sammalloomade positsioonist algsuuste (Protostomia) fülogeneesis. Limused, rõngussid ja käsijalgsed (+pärgussid) koos konuvaklade ja kärssussidega ilmselt moodustavad ühe klaadi, kuid kuhu paigutada mainitud rühmade suhtes sammalloomad? Halanych (2004) on selle hõimkonna paigutanud mitte-Ecdysozoa algsuuste sõsarrühmaks, mis automaatselt tähendab, et ka sellised hõimkonnad nagu lameussid ja keriloomad kuuluvad Lophotrochozoa hulka (vt. ülaltoodud joonist). Kuid näiteks Glenner et al. (2004) järgi võiks sammalloomad arvata klaadi, mis hõlmab ainult kärssusse, rõngusse, käsijalgseid, konuvaklasid ja limuseid (vt. joonist), ja seetõttu omandaks Lophotrochozoa hoopis kitsama tähenduse. Kui nii, siis võiks Ecdysozoa sõsarrühma nimeks olla Spiralia (vt. näiteks Valentine, 1997; Nielsen, 2001; Giribet, 2002). Cavalier-Smith (1998) moodustas keriloomade, ripskõhtsete, lõugsuude ja lameusside jaoks taksoni Platyzoa. Giribet (2002) on sinna lisanud ka Cycliophora ja Micrognathozoa, kusjuures Platyzoa on Lophotrochozoa sõsarrühmaks. Halanych (2004) on omakorda Platyzoa hulka lisanud veel kummarloomad ning seda taksonit käsitlenud Lophotrochozoa osana. Platyzoa monofüleetilisus on küsitav, kuid Cycliophora, kummarloomad ja keriloomad (tõenäoliselt ka Micrognathozoa) ilmselt on lähisugulased (e. g. Glenner et al., 2004; Telford et al., 2005). Ripskõhtsete, lameusside ja lõugsuude seos viimati mainitud rühmadega on ebaselge.
Käsnade (Demospongiae, Calcarea, Hexactinellida) fülogenees ülaltoodud joonisel on Müller (2001) järgi. Sellisele fülogeneesile leidsid Borchiellini et al. (2001) pisut kinnitust 18S rDNA järjestustest ja seda eelistas ka Halanych (2004). Enamjaolt kipub 18S rDNA ikkagi eelistama Silicea hüpoteesi (Demospongiae+Hexactinellida, kes siis on sõsarrühmaks Calcarea+Eumetazoa klaadile) (e. g. Peterson & Eernisse, 2001; Manuel et al., 2003; Eernisse & Peterson, 2004; Wallberg et al., 2004). Ka mõned paleontoloogid pooldavad pigem Silicea (=Silicispongea) hüpoteesi (Botting & Butterfield, 2005). Käsnade fülogeneesi teeb keerulisemaks veel see, et hiljuti on 18S ja 28S rDNA järjestuste põhjal päriskäsnade hulgast välja arvatud Homoscleromorpha esindajad (Borchiellini et al., 2004), kes võiksid mõnede tunnuste (akrosoom spermides ja IV tüüpi kollageeni sisaldava basaalmembraani olemasolu) järgi Calcarea asemel hoopis olla pärishulkraksete sõsarrühmaks. Borchiellini et al. (2004) poolt Homoscleromorpha välja arvamine päriskäsnadest ei pruugi väga vale olla, kuid võib siiski osutuda mittevajalikuks, kuna on selgunud, et Oscarella carmela (Homoscleromorpha) on mitokondriaalse DNA (Wang & Lavrov, 2007) ja rakutuuma valgugeeni (65 geeni; Jiménez-Guri et al., 2007) analüüside põhjal lähemal päriskäsnadele kui pärishulkraksetele. Wang & Lavrov (2007) ning Jiménez-Guri et al. (2007) analüüsides puudusid aga teised käsnad (lubi- ja klaaskäsnad), mistõttu Homoscleromorpha fülogeneetilise positsiooni kohta on veel vara lõplikku vastust anda. Kahe klaaskäsna peaaegu täielik mitokondri (mt) genoom on siiski juba sekveneeritud Haen et al. (2007) poolt, kuid nende tulemused muudavad olukorra hoopis veelgi keerulisemaks. Nimelt leidsid Haen et al. (2007), et üllataval kombel sarnaneb klaaskäsnade mt genoom väga kahekülgsete omaga; need sarnasused puudutavad järgmisi mtDNA tunnuseid: teatud muutused geneetilises koodis, rRNA ja tRNA molekulide sekundaarstruktuur, mt genoomi üldine organisatsioon ja kiire evolutsioon võrreldes teiste mittekahekülgsetega. Lisaks neile kvalitatiivsetele tunnustele toetavad klaaskäsnade ja kahekülgsete lähisugulust mõned fülogeneetilised analüüsid (joon. 4A Haen et al., 2007 töös), kuid rakendades keerukamat CAT mudelit1, leiti et klaaskäsnad on kõige lähemalt suguluses päriskäsnadega ja nende sõsarrühmaks on Oscarella (joon. 4B Haen et al., 2007 töös). Kahjuks ei olnud aga CAT mudelit rakendades klaaskäsnade fülogeneetiline positsioon stabiilne taksoni valimi suhtes (Haen et al., 2007). Selguse toomiseks käsnade ja üldse loomade varase fülogeneesi kohta, on vaja sekveneerida veel lubikäsnade ja kammloomade mt genoom ning hulgaliselt rakutuuma geene kammloomadelt, lubi- ja klaaskäsnadelt.
Naastloomi (Placozoa) pole joonisel toodud, kuna nende fülogeneetiline positsioon on üsna ebaselge. 18S rDNA järjestused viitavad sellele, et nad kuuluvad pärsihulkraksete hulka (e. g. Wallberg et al., 2004), kuid täpsemalt ei ole võimalik veel kindlaks teha. Ühe hüpoteesi järgi on Trichoplax adhaerens (ainuke kirjeldatud liik naastloomade hõimkonnas) sekundaarselt lihtsustunud kõrverakne. Selle hüpoteesi kontrollimiseks uurisid Ender & Schierwater (2003) 16S rDNA molekuli struktuuri ja mitokondri genoomi ning leidsid, et kõrveraksete hulka neid klassifitseerida ei saa. Väga ootamatule tulemusele jõudsid aga Dellaporta et al. (2006), kes sekveneerisid naastlooma Trichoplax adhaerens mitokondri (mt) genoomi. Nimelt selgus, et selle naastlooma mitokondri genoom (u. 43 000 bp) on üle kahe korra suurem kui see on tüüpiliselt loomadel (15 000-20 000 bp) ja see suurus ei ole ilmselt sekundaarset päritolu, kuna sarnaselt hulkraksete loomade lähisugulastele (seened, kaelusviburlased) on olemas intronid, intrageensed speisserid (intragenic spacers) ja mitmed valgugeenid, mille funktsioon teadmata. Kuigi Dellaporta et al. (2006) poolt tehtud valgugeenide fülogeneetilised analüüsid võibolla lõplikku vastust ei anna, näitab naastloomade selline mt genoomi struktuur, et neid ei saa hästi pärishulkraksete hulka arvata (nagu võiks arvata 18S rDNA järjestuste põhjal): paistab, et nad peaksid olema lahknenud teistest loomadest juba enne käsnasid ja seega võiks nende morfoloogiline lihtsus (sümmeetria puudumine, epiteeli aluse basaalmembraani puudumine, ainult neil somaatilist rakutüüpi) olla primaarne. Signorovitch et al. (2007) sekveneerisid lisaks veel kolme naastlooma (kes kõik esindavad ilmselt eri liike) mt genoomi ning selgus, et need on mõnevõrra väiksemad (u. 33 000-37 000 bp) kui Trichoplax adhaerens oma. Huvitav on siiski märkida, et kui Haen et al. (2007) kasutasid fülogeneesi rekonstrueerimisel (mtDNA põhjal) CAT mudelit (vt. ülal), paigutus Trichoplax kahekülgsete sõsarrühmaks, kuigi statistiline toetus Bilateria+Trichoplax klaadile ei olnud eriti tugev. Kui Trichoplax adhaernsi genoom saab sekveneeritud, siis võib loodetavasti kindlalt öelda, kuhu see kummaline organism loomariigis paigutada.
Molekulaarsed andmed on järjekindlalt (peamiselt ribosoomi väikse alaühiku geenide põhjal) pakkunud kahekülgsete (Bilateria) sõsarrühmaks kõrverakseid (Cnidaria), samas kui on olemas mitmeid morfoloogilisi tunnuseid (e. g. mesoderm), mis ühendavad kammloomi (Ctenophora) ja kahekülgseid (vt. Nielsen, 2001). Kuna kammloomade ja kahekülgsete närvisüsteem ja lihastik paistavad olevat keerulisemad võrreldes kõrveraksetega, siis on raske ette kujutada nii nende tunnuste sõltumatut kujunemist kammloomadel ja kahekülgsetel kui ka lihtsustumist pelaagilistel meduusidel (Podar et al., 2001). Wallberg et al. (2004) arutlesid sellel teemal ning leidsid, et käsitleda kõrverakseid kahekülgsete sõsarrühmana (nn. Planulozoa hüpotees) on täiesti mõeldav.
Limaeosloomi on üldiselt käsitletud protistidena, kuid alates 1990. aastate keskpaigast (e. g. Smothers et al., 1994) on selgeks saanud, et nad pärinevad hulkraksetest ja seega on nad sekundaarselt lihtsustunud. Mõnede morfoloogiliste tunnuste alusel võiks limaeosloomad (Myxozoa) arvata kõrveraksete klassi hüdraloomad (Hydrozoa): limaeosloomade polaarkapslid (polar capsules) sarnanevad väga kõrveraksete nematotsüstidega (organellid kõrverakkudes) ja neil on teatud sarnasusi (parasiitne eluviis) hüdraloomade perekonnaga Polypodium (vt. Kent et al., 2001). Limaeosloomade päritolu kõrveraksetest kahtlustas juba Ðtolc 1899. aastal (Kent et al., 2001 järgi). Enamik ribosoomi väikse alaühiku (18S) RNA molekuli analüüse aga paigutab limaeosloomad hoopis bilateraalsümmeetriliste loomade hulka või nende sõsarrühmaks (Smothers et al., 1994; Wallberg et al., 2004). Molekulaarsete andmete seast paistavad seda toetavat ka Anderson et al. (1998) tulemused, kes leidsid, et limaeosloomadel on olemas kahekülgsetele omased Hox geenid. Päritolu kahekülgsetest toetab ka avastus, et ussitaoline Buddenbrockia plumatellae (magevee sammalloomade kehaõõne parasiit) kuulub limaeosloomade hulka (Monteiro et al., 2002; Okamura et al., 2002): tal arenevad samasugused polaarkapslid nagu teistel limaeosloomadel ning tema usjas keha ja lihastik viitavad päritolule kahekülgsetest. Buddenbrockia plumatellae (klass Malacosporea) süstemaatiline kuuluvus oli kaua aega täielik mõistatus (vt. Okamura & Canning, 2003). Nüüd on aga selgunud, et limaeosloomad kuuluvad siiski kõrveraksete hulka: Jiménez-Guri et al. (2007) sekveneerisid 50 valgugeeni limaeoslooma Buddenbrockia rakutuumast ja näitasid, et see kuulub Medusozoa (hüdraloomad, täringmeduusid, karikmeduusilised ja karikloomad) klaadi. Samuti tehti kindlaks, et Anderson et al. (1998) poolt sekveneeritud Hox geenid pärinevad hoopis peremeestelt (sammalloom ja haug), keda uurimise all olnud limaeosloomad parasiteerivad, ja mitte limaeosloomadelt endalt (Jiménez-Guri et al., 2007). Jiménez-Guri et al. (2007) tulemuste põhjal tuleks ülaltoodud kladogrammi vastavalt muuta. Kui limaeosloomad kuuluvad Medusozoa klaadi, siis peaks neil olema lineaarne mitokondri genoom, mitte nagu teistel kõrveraksetel (st. õisloomadel) ja loomadel (ja üldse enamikel eukarüootidel), kelle mitokondritele on iseloomulik rõngaskromosoomi olemasolu (Bridge et al., 1992). Kahjuks pole aga veel midagi teada limaeosloomade mitokondri genoomist. Limaeosloomad täpsema fülogeneetilise positsiooni leidmine Medusozoa klaadis osutub ilmselt küllaltki keeruliseks, isegi kui taksonivalimit (klaadis Medusozoa) Jiménez-Guri et al. (2007) andmestikus oluliselt suurendada, kuna limaeosloomade ja hüdralooma Polypodium (potentsiaalselt limaeosloomade lähim sugulane) geenijärjestused on väga divergentsed võrreldes teiste loomadega (Siddall & Whiting, 1999; Kim et al., 1999; Collins, 2002; Jiménez-Guri et al., 2007).
Järgmised saladusliku päritoluga loomad on vaheloomad - Orthonectida ja Rhombozoa. Kas neid saab ühendada ühte rühma (klaadi) Mesozoa, mida tihti tehakse, või mitte? Kus nad asuvad hulkraksete loomade fülogeneesis? Molekulaarsed uuringud näitavad, et nad on kahekülgsed (Katayama et al., 1995; Hanelt et al., 1996; Pawlowski et al., 1996), kuid täpsemalt midagi kindlat öelda veel ei saa. Nad võivad olla näiteks tugevalt degenereerunud lameussid. Zrzavy (2001) arvab, et Mesozoa on siiski monofüleetiline rühm, mis võib lähedalt seotud olla umbeliselaadsetega (Acoelomorpha). Vilasktomploomade (Rhombozoa: Dicyemida) päritolu kohta on olemas väga huvitav hüpotees. Noto & Endoh (2004) pakkusid välja, et need peajalgsete obligaatsed parasiidid on kimäärid mingist protistist (võimalik et ripsloomast) ja nende peremehest. Sellised kimäärid võivad olla kujunenud mõnest ainuraksest protistist, kes on horisontaalse geeniülekande teel omandanud oma peremehelt geene, mis võisid põhjustada ka hulkraksuse tekke (Noto & Endoh, 2004). Näiteks 18S rRNA (e. g. Hanelt et al., 1996) ja Hox geenid (Kobayashi et al., 1999) viitavad sellele, et nad on kahekülgsed, võibolla isegi Lophotrochozoa klaadi esindajad. Kuid nagu Noto & Endoh (2004) märgivad, on nad morfoloogiliselt pigem protistid kui loomad. Sarnasusele protistidega viitavad näiteks järgmised (primitiivsed) tunnused: äärmiselt lihtsustunud keha ehitus (nad koosnevad 20-40 rakust), mitokondrite tubulaarsed kristad (loomadel on need lamedad), endotsütoosi võime keha välispinnal, ekstratsellulaarse maatriksi puudumine (ka umbeliselaadsetel see puudub). Mingid geenid võiksid tõenäoliselt horisontaalselt üle kanduda, kuid kas on võimalik, et asendub selline oluline geen nagu 18S rRNA? Vilasktomploomade genoomi või enamate geenide sekveneerimisel oleks võimalik kindlaks teha, kellega on tegemist. Tõenäoliselt piisaks mitokondri genoomist, kuna on väga vähe tõenäoline (või suisa võimatu?), et see asenduks horisontaalsel ülekandel mõne teise organismi omaga.
Rühmasid Acoela ja Nemertodermatida (liidetakse Acoelomorpha alla) käsitletakse tavaliselt lameussidena (Tyler, 2003). Viimase aja molekulaarsed uuringud on aga selle kahtluse alla seadnud (e. g. Ruiz-Trillo et al., 1999; 2002; 2004; Telford et al., 2003). Nimelt on nende uuringutega näidatud, et umbelised (Acoela) ja Nemertodermatida (Ruiz-Trillo et al., 2002; Telford et al., 2003) pole mitte lameussid, vaid on lahknenud enne kõiki teisi bilateraalsümmeetrilisi loomi. Kahtluse alla on seatud ka see, et Acoela ja Nemertodermatida on sõsarrühmad, mis tundub neid "lameusse" uurivatele teadlastele täiesti absurdsena (Tyler, 2003). Kuna mitmed statistilised testid Ruiz-Trillo et al. (2002) ja Telford et al. (2003) töödes ei lükanud täielikult ümber Acoelomorpha hüpoteesi ja kuna need kaks rühma on erakordselt sarnased, siis tegid Telford ja kolleegid (2003) ettepaneku käsitleda taksonit Acoelomorpha eraldi hõimkonnana. Hoopis huvitavale tulemusele jõudsid Philippe et al. (2007), kellel olid fülogeneetiliste analüüside tarvis olemas 68 valgugeeni järjestust umbeliselt Convoluta pulchra: selgus, et umbelised on kõige tõenäolisemalt teissuused (Deuterostomia), võib-olla Xenoturbella sõsarrühmaks. Tasub mainida, et teatud epidermise ripsmete (cilia) ultrastruktuuri tunnused viitavad Xenoturbellida ja Acoelomorpha lähisugulusele (Franzén & Afzelius, 1987; Lundin, 1998); neid sarnasusi olen ma pidanud plesiomorfseteks tunnusteks. Philippe et al. (2007) tulemused ei anna siiski lõplikku vastust, kuna analüüsitud umbelise järjestused olid väga divergentsed (evolutsioneeruvad kiiresti) võrreldes teiste loomadega ning seetõttu on korrektse fülogeneetilise positsiooni leidmine keeruline (seda mainisid ka autorid). Umbeliste päritolule teissuustest räägivad vastu (kuid on kooskõlas eelnevalt viidatud töödega) aga Sempere et al. (2006) tulemused (vt. ka Pasquinelli et al., 2003), kellel ei õnnestunud ühelt uuritud umbeliselt (Childia sp.) leida ühtegi teissuustele (ega ka algsuustele) omast regulaator RNA (microRNA=miRNA) molekuli. Sempere et al. (2006) järgi puuduvad käsnadel miRNA molekulid üldse, kõrveraksetel on neid 2 ning algsuuste ja teissuuste (=Nephrozoa) eellasel oli neid olemas vähemalt 20. 16 miRNA molekulist, mis uuritud umbelisel leiti, olid ainult 6 miRNA-d ühised Nephrozoa esindajatega (Sempere et al., 2006) ning seetõttu võiks arvata, et umbelised on lahknenud enne ülejäänud kahekülgseid (st. ei kuulu Nephrozoa klaadi). Sempere et al. (2006) töö puuduseks võib pidada seda, et mitmeid olulisi taksoneid (Nemertodermatida, Xenoturbellida) ei oldud analüüsitud: väga huvitav oleks teada, kas Xenoturbella esindajatel on umbelistega sarnane miRNA-de komplekt või on neil siiski olemas teissuustele omased miRNA-d [kuna paistab, et nad tõepoolest teissuuste hulka kuuluvad (Bourlat et al., 2006; Philippe et al., 2007)].
Probleemsed taksonid on veel harjaslõugsed, sammalloomad ja käsijalgsed (+pärgussid) kuna ei teata, kas neid paigutada teissuuste (Deuterostomia) või ürgsuuste (Protostomia) hulka. Kuna nad on oma tunnustekomplekti poolest nende kahe rühma vahepealsed, siis on zooloogid palju nende hõimkondade kuuluvuse üle vaielnud. Molekulaarsete järjestuste analüüsid (18S rRNA) kipuvad neid kõiki rühmi paigutama ürgsuuste hulka (Adoutte et. al., 2000). Sammalloomade ja käsijalgsete kuuluvust ürgsuuste hulka (täpsemalt Lophotrochozoa klaadi) toetavad peale 18S rRNA geenide veel mitokondri genoomi analüüsid ja Hox geenid (e. g. de Rosa, 2001, Larget et al., 2005, Passamaneck & Halanych, 2004). Ka harjaslõugsed on üsna kindlalt ürgsuused, kuid nende täpsem positsioon on veel ebaselge (Papillon et al., 2004; Helfenbein et al., 2004; Marlétaz et al., 2006; Matus et al., 2006).
Tahaks veel rääkida ümarussidest, täpsemalt ühest varbussist (Caenorhabditis elegans), kelle genoom on sekveneeritud.Tänapäeval on kaks peamist teooriat lülijalgsete põlvnemise kohta: nad kas on lähedalt suguluses rõngussidega (Nielsen, 2001; joon. 1) või hoopis ümarussidega (Aguinaldo et al., 1997). Kuid mõlema teooria kohaselt on nad fülogeneetiliselt üksteisele lähemal (kuuluvad Protostomia klaadi), kui kumbki on teissuustele (Deuterostomia). Juba mõnda aega on olemas järgmiste organismide täielikud genoomi järjestused: Drosophila (äädikakärbes), Caenorhabditis (varbuss) ja Homo (inimene). Kaks esimest (lülijalgne ja ümaruss) on algsuused (Protostomia) ning viimane (keelikloom) on deuterostoom. Sellest lähtuvalt võiks nagu arvata, et nende kolme organismi genoomi analüüsimine klasterdab lülijalgsed ja ümarussid ühte ja jätab keelikloomad omaette. Tundub, et asi pole üldsegi mitte nii. Nimelt, Ecdysozoa hüpoteesi (Aguinaldo et al., 1997) kontrollimiseks otsisid Blair et al. (2002) andmebaasidest üle 100 ortoloogse geeni ning viisid läbi fülogeneetilised analüüsid. Üllatuseks selgus, et lülijalgsed on inimesele lähemad sugulased kui nad on ümarussidele, kinnitades hoopis varasemaid teooriaid (20. sajandi esimene pool), kus ilma kehaõõneta (acoelomata; e. g. lameussid), esmase kehaõõnega (pseudocoelomata; e. g. ümarussid) ja teisese kehaõõnega (coelomata2; e. g. rõngussid, lülijalgsed, keelikloomad) loomi käsitleti erinevate evolutsiooni harudena. Sellistele tulemustele on jõudnud ka teised varasemad uuringud (kuigi nende tööde eesmärk ei olnud kontrollida Ecdysozoa hüpoteesi), kus on kasutatud paljusid valke kodeerivaid tuumageene (Wang et al., 1999; Hausdorf, 2000). Seda kinnitavad ka Wolf et al. (2004), kes kasutasid fülogeneetilistes analüüsides juba 500 ortoloogset proteiini! Kuidas seda kõike seletada? Kas tõesti võib ümarussi Caenorhabditis elegans selline positsioon olla artefakt, mis paneks kahtluse alla molekulaarse fülogeneesi meetodite usaldusväärsuse? Või hoopis lihtsalt ei teata loomade fülogeneesist tõepoolest ikka veel peaaegu mitte midagi, erinevalt sellest mida viimasel ajal väidetakse (Adoutte et al., 2000; Nielsen, 2001; Giribet, 2002). Üsna võimalik, et ümarussi C. elegans selline positsioon ongi artefakt, kuid see ei pea tingimata tähendama, et molekulide primaarjärjestuse analüüse ei tasu üldse usaldada. Probleemiks võib olla taksonite (väike) valim. Mingil põhjusel (näiteks kui lisatud vähesed taksonid lahknesid üksteisest väga ammu) võib saadud fülogeneesi puu siiski vale olla ning järjestuste hulga suurendamine lihtsalt võimendaks viga (Hillis et al., 2003; vt. ka kommentaare imetajate fülogeneesi leheküljel). Antud probleemi lahendamiseks (Coelomata versus Ecdysozoa) ei piisa andmestiku pidevast suurendamisest nende väheste taksonite jaoks (Drosophila, Caenorhabditis, Homo), vaid analüüsidesse tuleb lisada uusi taksoneid. Mis puutub järjestuste analüüsi, siis on Wolf et al. (2004) töö praktiliselt identne Blair et al. (2002) omaga, kuna alates mingist järjestuse pikkusest pole enam oluline palju geene sul analüüsis on, ühele tulemusele jõuab ükskõik milline fülogeneesi rekonstrueerimise meetod nii või teisiti (Hillis et al., 2003). Kuid peale molekulide järjestuse analüüsi kasutasid Wolf et al. (2004) evolutsiooniliste suhete leidmiseks nende kolme mudelorganismi vahel veel näiteks insertsioonide/deletsioonide (indels) esinemist valkudes ja erinevate ortoloogsete geenide esinemise mustreid (eelduseks on, et need analüüsitavatest organismides, kes omavad kõige rohkem ühiseid geene, on tõenäoliselt lähimad sugulased). Kõikidel juhtudel paistsid putukad rohkem sarnased olevat inimesele kui ümarussidele. Nende viimaste argumentide vastuväiteks on Copley et al. (2004) töö, kes C. elegans genoomi eripärasid3 arvesse võttes leidsid pisut toetust Ecdysozoa hüpoteesile. Enamik zoolooge pooldab siiski kahekülgsete jagunemist alg- ja teissuusteks (mitte rühmadeks acoelomata, pseudocoelomata, coelomata), kusjuures üha populaarsemaks on muutumas Ecdysozoa hüpotees, mis võib ikkagi õigeks osutuda [näiteks väga huvitavatele tulemustele jõudsid Haase et al. (2001), kes näitasid, et mädarõika peroksidaasi (horseradish peroxidase) vastane immuunreaktiivsus kesknärvisüsteemis esineb ainult Ecdysozoa klaadi kuuluvate hõimkondade esindajatel]. Seega on kokkuvõtteks põhjust kahelda Blair et al. (2002) ja teiste sarnasete tööde tulemustes ning seetõttu pole ma ülal toodud kladogrammil neid arvesse võtnud. Loomade fülogeneesi väljaselgitamisel tuleb eeskuju võtta imetajate fülogeneesi uuringutest - kasutada tuleb palju taksoneid ja palju geene (häid tulemusi annab juba 10-20 geeni analüüsimine). Hiljuti avaldatud töös demonstreerisid Philippe et al. (2004) just taksoni valimi mõju fülogeneesi rekonstrueerimisele. Nad analüüsisid üle saja geeni ja 36 eukarüooti, kusjuures suur osa liikidest olid loomad (kolm keeliklooma, neli lülijalgset, seitse ümarussi ja üks lameuss). Lülijalgsed, lameuss ja ümarussid klasterdusid ühte klaadi erinevalt teistest genoomi tasemel tehtud analüüsidest, kus taksoni valim oli märgatavalt väiksem (e. g. Blair et al., 2002; Wolf et al., 2004). Kuid Ecdysozoa hüpotees ei leidnud kinnitust, kuna ümarusside sõsarrühmaks oli hoopis lameuss mitte lülijalgsed. Nagu ka artikli autorid mainisid võis selle põhjuseks olla jälle nn. long-branch attraction (lameuss ja ümarussid paistsid tunduvalt kiiremini evolutsioneeruvat kui teised) ning selle efekti leevendamiseks tuleks analüüsi võtta esindajaid ka teistest hõimkondadest. Philippe ja kolleegid (2005) paistavad olevat lahendanud ka selle probleemi. Nad täiendasid ja modifitseerisid eelmist andmekogumit (Philippe et al., 2004) ja demonstreerisid väga veenvalt Ecdysozoa, Lophotrochozoa ja Protostomia klaadide monofüleetilisust ja seda kui oluliselt võib mõjutada fülogeneesi rekonstrueerimist taksoni valim. Lameusside ja ümarusside grupeerumine oli tõepoolest artefakt. Nende kahe hõimkonna õiget fülogeneetilise positsiooni leidmist takistasid samad probleemid, mis on ette tulnud 18S rRNA geenide kasutamisel (vt. Aguinaldo et al., 1997). Kuigi erinevate loomahõimkondade arv oli Philippe et al. (2005) töös väga väike (kõrverakne, kammloom, keelikloomad, okasnahkne, loimur, ümarussid, lülijalgsed, lameussid, mollusk, rõnguss) võivad 18S rRNA geenide põhjal identifitseeritud Ecdysozoa (Aguinaldo et al., 1997) ja Lophotrochozoa (Halanych et al., 1995) klaadid osutuda tõelisteks. Samas üritavad Philip et al. (2005) näidata Ecdysozoa (ja Opisthokonta) mittemonofüleetilisust, kuid nende analüüsid piirduvad jällegi nende üksikute mudelorganismidega. Enamik genoomi tasemel, ainult üksikuid mudelorganisme hõlmavad, tehtud analüüsid (Mushegian et al., 1998; Blair et al., 2002; Wolf et al., 2004; Dopazo et al., 2004; Philip et al., 2005) toetavad Coelomata hüpoteesi. Dopazo & Dopazo (2005) leidsid aga, erinevalt eelnevatele töödele, tõendeid lülijalgsete-ümarusside klaad eksisteerimise kohta. Sellistele tulemustele jõudsid Dopazo & Dopazo (2005) siis kui nad olid välja valinud sellised geenid, mis evolutsioneerusid sarnaselt nii lülijalgsetel kui ümarussil, või eemaldasid analüüsidest ümarussi kiiresti evolutsioneeruvad geenid.
Põgus stsenaarium loomade (õigemini kahekülgsete) varasest evolutsioonist. Tihti peetakse lameusse kõige primitiivsemateks kahekülgseteks. Arvestades umbeliselaadsete (keda tavaliselt lameussidena käsitletaksegi) fülogeneetilist positsiooni, võib see ka nii olla. Oletame, et eelkambriumis (>543 miljonit aastat tagasi) olidki enamus kahekülgsetest loomadest lameusside (ripsusside) taolised. Hiljem, Vara-Kambriumis, toimus nende lameusside adaptiivne radiatsioon (nagu platsentaalide evolutsioonis, vt. Imetajate fülogenees), mida nimetatakse Kambriumi plahvatuseks (Cambrian explosion), kuna siis ilmusid lühikese ajaga (geoloogilises ajaskaalas) enamus tänapäeva loomahõimkondadest. Selles evolutsioonilises mitmekesistumises kujunesid erinevatest ripsussidest mitmesuguste ehitusplaanidega loomad, mida tänapäeval klassifitseerime erinevateks hõimkondadeks. Sellest tulenevalt on seletatav ka tänapäeva erinevate "lameusside" [Platyhelminthes (lameussid kitsas mõttes), Xenoturbellida, Acoela ja Nemertodermatida (umbeliselaadsed)] hajutatud paiknemine loomade fülogeneesis. Kahjuks puuduvad paleontoloogilised leiud (või pole neid osatud veel õigesti tõlgendada) selle kohta, et Kambriumi plahvatuses tekkinud hõimkondadele eelnesid lameussid. Erinevalt selgroogsetest loomadest, ei jäta ainult pehmetest kudedest koosnevad loomad fossiilidena nii hästi jälgi. Olid nendeks Kambriumi fossiilide eellasteks mainitud hüpoteetilised "lameussid" või midagi muud, pidid nad ilmselt olema üsna väiksed, lihtsa ehituse ja säilima fossiilidena halvasti. Joonisel toodud umbeliselaadsete fülogeneetiline positsioon lisab pisut toetust sellele, et nendeks võisid lameussid olla. On täiesti mõeldav, et kompleksne morfoloogia (e. g. segmentatsioon, silmad ning muud keerulisemad elundid ja elundkonnad) kujunes erinevates ripsusside rühmades välja sõltumatult, kuna on leitud, et põhiosa morfoloogilise keerukuse eest vastutavast geneetilisest aparatuurist oli tõenäoliselt olemas juba kõrveraksetel (vt. näiteks Miller et al., 2005).
1 - Biokeemiliste omaduste tõttu võib valgujärjestuse teatud positsioonis tavaliselt leida ainult kahte kuni nelja erinevat aminohapet (kuigi aminohappeid on kokku 20); millised need konkreetsed aminohapped olla võivad, sõltub aga sellest, millise positsiooniga on tegemist, st. et tavaliselt on igale positsioonile omane teatud kitsas aminohapete profiil. Seda biokeemilist reaalsust ei võtta hästi arvesse standardsed empiirilised aminohapete asendusi kirjeldavad maatriksid (nt. WAG), kus igale positsioonile antakse samasugune lai aminohapete profiil. CAT mudelis aga võetakse arvesse positsiooni-spetsiifilisi aminohapete asenduse protsesse ja enamikes positsioonides antakse suur esinemise tõenäosus ainult teatud aminohapetele (Lartillot & Philippe, 2004; Lartillot et al., 2007).
2 - teisese kehaõõnega loomad olid jaotatud algsuusteks (Protostomia) ja teissuusteks (Deuterostomia). Algsuuste tähendus on tänapäeval muutunud, kuna sinna paigutatakse ka kehaõõneta ja esmase kehaõõnega loomahõimkonnad.
3 - Caenorhabditis elegans (võibolla ka paljude teiste ümarusside) genoom on tõenäoliselt sekundaarselt lihtsustunud - evolutsiooni jooksul on kadunud palju suurem hulk geene võrreldes selgroogsete ja putukatega.
Viited
Adoutte, A., G. Balavoine, N. Lartillot, O. Lespinet, B. Prud'homme, & R. de Rosa. 2000. The new animal phylogeny: Reliability and implications. Proceedings of the National Academy of Sciences. USA 97: 4453–4456. [pdf]
Aguinaldo, A. M., Turbeville, J. M., Linford, L. S., Rivera, M. C., Garey, J. R., Raff, R. A., Lake, J. A. 1997. Evidence for a clade of nematodes, arthropods and other moulting animals. Nature 387: 489–493.
Anderson, C. L., Canning, E. U., Okamura, B. 1998. A triploblast origin for Myxozoa? Nature 392: 346–347.
Blair, J. E., Ikeo, K., Gojobori, T. & Hedges, S. B. 2002. The evolutionary position of nematodes. BMC Evolutionary Biology 2: 7. [pdf]
Borchiellini, C., Manuel, M., Alivon, E., Boury-Esnault, N., Vacelet, J. & Le Parco, Y. 2001. Sponge paraphyly and the origin of Metazoa. Journal of Evolutionary Biology 14: 171–179.
Borchiellini, C., Chombard, C., Manuel, M., Alivon, E., Vacelet, J. & Boury-Esnault, N. 2004. Molecular phylogeny of Demospongiae: implications for classification and scenarios of character evolution. Molecular Phylogenetics and Evolution 32: 823–837.
Botting, J. P & Butterfield, N. J. 2005. Reconstructing early sponge relationships by using the Burgess Shale fossil Eiffelia globosa, Walcott. Proceedings of National Academy of Science. USA 102: 1554–1559. [pdf]
Bourlat, S. J., Juliusdottir, T., Lowe, C. J., Freeman, R., Aronowicz, J., Kirschner, M., Lander, E. S., Thorndyke, M., Nakano, H., Kohn, A. B., Heyland, A., Moroz, L. L., Copley, R. R. & Telford, M. J. 2006. Deuterostome phylogeny reveals monophyletic chordates and the new phylum Xenoturbellida. Nature 444: 85–88.
Bridge, D., Cunningham, C. W., Schierwater, B., DeSalle, R. & Buss, L. W. 1992. Class-level relationships in the phylum Cnidaria: Evidence from mitochondrial genome structure. Proceedings of the National Academy of Sciences. USA 89: 8750–8753. [pdf]
Cavalier-Smith, T. 1998. A Revised Six-Kingdom System of Life. Biological Reviews 73: 203–266.
Collins, A. G. 2002. Phylogeny of Medusozoa and the evolution of cnidarian life cycles. Journal of Evolutionary Biology 15: 418–432.
Copley, R. R., Aloy, P., Russell, R. B. & Telford, M. J. 2004. Systematic searches for molecular synapomorphies in model metazoan genomes give some support for Ecdysozoa after accounting for the idiosyncrasies of Caenorhabditis elegans. Evolution & Development 6: 164–169.
Dellaporta, S. L., Anthony Xu, A., Sagasser, S., Jakob, W., Moreno, M. A., Buss, L. W. & Schierwater, B. 2006. Mitochondrial genome of Trichoplax adhaerens supports Placozoa as the basal lower metazoan phylum. Proceedings of the National Academy of Sciences. USA 103: 8751–8756. [pdf]
Dopazo H., Santoyo, J. & Dopazo, J. 2004. Phylogenomics and the number of characters required for obtaining an accurate phylogeny of eukaryote model species. Bioinformatics 20 (Suppl. 1): i116–i121. [pdf]
Dopazo, H. & Dopazo, J. 2005. Genome-scale evidence of the nematode-arthropod clade. Genome Biology 6: R41. [pdf]
Eernisse, D. J. & Peterson, K. J. 2004. The history of animals. 197-208 in Cracraft, J. & Donoghue, M. J. (eds.) Assembling the Tree of Life. Oxford University Press, New York. 592 pp.
Ender, A. & Schierwater, B. 2003. Placozoa are not derived cnidarians: Evidence from molecular morphology. Molecular Biology & Evolution 20: 130–134. [pdf]
Franzén, A. & Afzelius, B. A. 1987. The ciliated epidermis of Xenoturbella bocki (Platyhelminthes, Xenoturbellida) with some phylogenetic considerations. Zoologica Scripta 16: 9–17.
Giribet, G. 2002. Current advances in the phylogenetic reconstruction of metazoan evolution. A new paradigm for the Cambrian explosion? Molecular Phylogenetics and Evolution 24: 345–357.
Glenner, H., Hansen, A. J., S¸rensen, M. V., Ronquist, F., Huelsenbeck, J. P. & Willerslev, E. 2004. Bayesian Inference of the Metazoan Phylogeny: A Combined Molecular and Morphological Approach. Current Biology 14: 1644–1649; Erratum: Current Biology 15: 392–393 (2005).
Haase, A., Stern, M., Wächtler, K. & Bicker, G. 2001. A tissue-specific marker of Ecdysozoa. Development Genes and Evolution 211: 428–433.
Haen, K. M., Lang, B. F., Pomponi, S. A. & Lavrov, D. V. 2007. Glass Sponges and Bilaterian Animals Share Derived Mitochondrial Genomic Features: A Common Ancestry or Parallel Evolution? Molecular Biology & Evolution 24: 1518–1527. [pdf]
Halanych, K. M., Bacheller, J. D., Aguinaldo, A. M., Liva, S. M., Hillis, D. M. & Lake, J. A. 1995. Evidence from 18S ribosomal DNA that the lophophorates are protostome animals. Science 267: 1641–1643.
Halanych, K. M. 2004. The new view of animal phylogeny. Annual Review of Ecology, Evolution, and Systematics 35: 229–256.
Hanelt, B., van
Schyndel, D., Adema, C. M., Lewis, L. A. & Loker, E. S. 1996. The
phylogenetic position of Rhopalura ophiocomae (Orthonectida) based on
18S ribosomal DNA sequence analysis
Molecular Biology and Evolution 13: 1187–1191. [pdf]
Hausdorf, B. 2000. Early evolution of the Bilateria. Systematic Biology 49: 130–142.
Helfenbein, K. G., Fourcade, H. M., Vanjani, R. G. & Boore, J. L. 2004. The mitochondrial genome of Paraspadella gotoi is highly reduced and reveals that chaetognaths are a sister group to protostomes. Proceedings of National Academy of Science. USA 101: 10639–10643. [pdf]
Hillis, D. M., Pollock, D. D., McGuire, J.A. & Zwickl, D. J. 2003. Is sparse taxon sampling a problem for phylogenetic inference? Systematic Biology 52: 124–126.
Jiménez-Guri, E., Philippe, H., Okamura, B. & Holland, P. W. H. 2007. Buddenbrockia Is a Cnidarian Worm. Science 317: 116–118.
Katayama, T., Wada, H., Furuya, H., Satoh, N. & Yamamoto, M. 1995. Phylogenetic Position of the Dicyemid Mesozoa Inferred from 18S rDNA Sequences. The Biological Bulletin 189: 81–90. [pdf]
Kent, M. L., K. B. Andree, J. L. Bartholomew, M. El-Matbouli, S. S. Desser, R. H. Devlin, S. W. Feist, R. P. Hedrick, R. W. Hoffmann, J. Khattra, S. L. Hallett, R. J. G. Lester, M. Longshaw, O. Palenzeula, M. E. Siddall, and C. Xiao. 2001. Recent advances in our knowledge of the Myxozoa. Journal of Eukaryotic Microbiology 48: 395–413.
Kim, J., Kim, W. & Cunningham, C. W. 1999. A New Perspective on Lower Metazoan Relationships from 18S rDNA Sequences. Molecular Biology and Evolution 16: 423–427. [pdf]
Kobayashi, M., Furuya, H. & Holland, P. W. H. 1999. Dicyemids are higher animals. Nature 401: 762.
Larget, B., Simon, D. L., Kadane, J. B. & Sweet, D. 2005. A Bayesian Analysis of Metazoan Mitochondrial Genome Arrangements. Molecular Biology and Evolution 22: 486-495. [pdf]
Lartillot, N. & Philippe, H. 2004. A Bayesian Mixture Model for Across-Site Heterogeneities in the Amino-Acid Replacement Process. Molecular Biology and Evolution 21: 1095–1109. [pdf]
Lartillot, N., Brinkmann, H. & Philippe, H. 2007. Suppression of long-branch attraction artefacts in the animal phylogeny using a site-heterogeneous model. BMC Evolutionary Biology 7(Suppl 1): S4. [pdf]
Lundin, K. 1998. The epidermal ciliary rootlets of Xenoturbella bocki (Xenoturbellida) revisited: new support for a possible kinship with the Acoelomorpha (Platyhelminthes). Zoologica Scripta 27: 263–270.
Manuel, M., Borchiellini, C., Alivon, E., Le Parco, Y., Vacelet, J., Boury-Esnault, N. 2003. Phylogeny and evolution of calcareous sponges : monophyly of calcinea and calcaronea, high level of morphological homoplasy, and the primitive nature of axial symmetry. Systematic Biology 52: 311–333.
Marlétaz, F., Martin, E., Perez, Y., Papillon, D., Caubit, X., Lowe, C. J., Freeman, B., Fasano, L., Dossat, C., Wincker, P., Weissenbach, J. & Le Parco, Y. 2006. Chaetognath phylogenomics: a protostome with deuterostome-like development. Current Biology 16: R577–R578.
Matus, D. Q., Copley, R. R., Dunn, C. W., Hejnol, A., Eccleston, H., Halanych, K. M., Martindale, M. Q. & Telford, M. J. 2006. Broad taxon and gene sampling indicate that chaetognaths are protostomes. Current Biology 16: R575–R576.
Miller, D. J., Ball, E. E. & Technau, U. 2005. Cnidarians and ancestral genetic complexity in the animal kingdom. Trends in Genetics 21: 536–539.
Monteiro, A. S., Okamura, B. & Holland, P. W. H. 2002. Orphan Worm Finds a Home: Buddenbrockia is a Myxozoan. Molecular Biology and Evolution 19: 968–971. [pdf]
Mushegian, A. R., Garey, J. R., Martin, J. & Liu, L. X. 1998. Large-Scale Taxonomic Profiling of Eukaryotic Model Organisms: A Comparison of Orthologous Proteins Encoded by the Human, Fly, Nematode, and Yeast Genomes. Genome Research 8: 590–598. [pdf]
Müller, W. E. G. 2001. Review: How was metazoan threshold crossed? The hypothetical Urmetazoa. Comparative Biochemistry and Physiology Part A 129: 433–460.
Nielsen, C. 2001. Animal Evolution: Interrelationships of the Living Phyla. Second Edition. Oxford University Press, Oxford. 568 pp.
Noto, T. & Endoh, H. 2004. A "chimera" theory on the origin of dicyemid mesozoans: evolution driven by frequent lateral gene transfer from host to parasite. BioSystems 73: 73–83.
Okamura, B., Curry, A., Wood, T. S. & Canning, E. U. 2002. Ultrastructure of Buddenbrockia identifies it as a myxozoan and verifies the bilaterian origin of the Myxozoa. Parasitology 124: 215–223.
Okamura, B. & Canning, E. U. 2003. Orphan worms and homeless parasites enhance bilaterian diversity. Trends in Ecology & Evolution 18: 633–639.
Papillon, D., Perez, Y., Caubit, X. & Le Parco, Y. 2004. Identification of Chaetognaths as Protostomes Is Supported by the Analysis of Their Mitochondrial Genome. Molecular Biology and Evolution 21: 2122–2129. [pdf]
Pasquinelli, A. E., McCoy, A., Jiménez, E., Saló, E., Ruvkun, G., Martindale, M. Q. & Baguñà, J. 2003. Expression of the 22 nucleotide let-7 heterochronic RNA throughout the Metazoa: a role in life history evolution? Evolution & Development 5: 372–378.
Passamaneck, Y. J. & Halanych, K. M. 2004. Evidence from Hox genes that bryozoans are lophotrochozoans. Evolution & Development 6: 275–281.
Pawlowski, J., Montoya-Burgos, J. I., Fahrni, J. F., Wuest, J. & Zaninetti, L. 1996. Origin of the Mesozoa inferred from 18S rRNA gene sequences. Molecular Biology and Evolution 13: 1128–1132. [pdf]
Peterson, K. J. & Eernisse, D. J. 2001. Animal phylogeny and the ancestry of bilaterians: inferences from morphology and 18S rDNA gene sequences. Evolution & Development 3: 170–205.
Philip, G. K., Creevey, C. J. & McInerney, J. O. 2005. The Opisthokonta and the Ecdysozoa may not be Clades: Stronger Support for the Grouping of Plant and Animal than for Animal and Fungi and Stronger Support for the Coelomata than Ecdysozoa. Molecular Biology and Evolution 22: 1175–1184. [pdf]
Philippe, H., Snell, E. A., Bapteste, E., Lopez, P., Holland, P. W. H. & Casane, D. 2004. Phylogenomics of Eukaryotes: Impact of Missing Data on Large Alignments. Molecular Biology and Evolution 21: 1740–1752. [pdf]
Philippe, H., Lartillot, N. & Brinkmann, H. 2005. Multigene Analyses of Bilaterian Animals Corroborate the Monophyly of Ecdysozoa, Lophotrochozoa and Protostomia. Molecular Biology and Evolution 22: 1246–1253. [pdf]
Philippe, H., Brinkmann, H., Martinez, P., Riutort, M. & Baguñà, J. 2007. Acoel Flatworms Are Not Platyhelminthes: Evidence from Phylogenomics. PLoS ONE 2: e717. [pdf]
Podar, M., Haddock, S. H. D., Sogin, M. L. & Harbison, G. R. 2001. A Molecular Phylogenetic Framework for the Phylum Ctenophora Using 18S rRNA Genes. Molecular Phylogenetics and Evolution 21: 218–230.
de Rosa, R. 2001. Molecular data indicate the protostome affinity of brachiopods. Systematic Biology 50: 848–859.
Ruiz-Trillo, I., Riutort, M., Littlewood, D. T. J., Hernieu, E. A., Baguñà, J. 1999. Acoel flatworms: earliest extant bilaterian metazoans, not members of Platyhelminthes. Science 283: 1919–1923.
Ruiz-Trillo, I., Paps, J., Loukota, M., Ribera, C., Jondelius, U., Baguñà, J., Riutort, M. 2002. A phylogenetic analysis of myosin heavy chain type II sequences corroborates that Acoela and Nemertoderematida are basal bilaterians. Proceedings of National Academy of Science. USA 99: 11246–11251. [pdf]
Ruiz-Trillo, I., Riutort, M., Matthew Fourcade, H., Baguñà, J. & Boore, J. L. 2004. Mitochondrial genome data support the basal position of Acoelomorpha and the polyphyly of the Platyhelminthes. Molecular Phylogenetics and Evolution 33: 321–332.
Sempere, L F., Cole, C. N., McPeek, M. A. & Peterson, K. J. 2006. The Phylogenetic Distribution of Metazoan microRNAs: Insights into Evolutionary Complexity and Constraint. Journal of Experimental Zoology (Mol Dev Evol) 306B: 575–588.
Siddall, M. E. & Whiting, M. F. 1999. Long-Branch Abstractions. Cladistics 15: 9–24.
Signorovitch, A. Y., Buss, L. W. & Dellaporta, S. L. 2007. Comparative Genomics of Large Mitochondria in Placozoans. PLoS Genetics 3: e13. [pdf]
Smothers, J. F., von Dohlen, C. D., Smith Jr., L. H. & Spall, R. D. 1994. Molecular Evidence That the Myxozoan Protists Are Metazoans. Science 265: 1719–1721.
Zrzavy, J. 2001. The interrelationships of metazoan parasites: a review of phylum- and higher-level hypotheses from recent morphological and molecular phylogenetic analyses. Folia Parasitologica 48: 81–103.
Telford, M. J., Lockyer, A. E., Cartwright-Finch, C., Littlewood, D. T. J. 2003. Combined large and small subunit ribosomal RNA phylogenies support a basal position of the acoelomorph flatworms. Proceedings of Royal Society of London B 270: 1077–1083.
Telford, M. J., Wise, M. J. & Gowri-Shankar, V. 2005. Consideration of RNA Secondary Structure Significantly Improves Likelihood-Based Estimates of Phylogeny: Examples from the Bilateria. Molecular Biology and Evolution 22: 1129–1136. [pdf]
Tyler, S. 2003. Platyhelminthes: the nature of a controversial phylum. http://devbio.umesci.maine.edu/styler/globalworming/platyhelm.htm
Valentine, J. W. 1997. Cleavage patterns and the topology of the metazoan tree of life. 94: Proceedings of National Academy of Science. USA 8001–8005. [pdf]
Wallberg, A., Thollesson, M., Farris, J. S. & Jondelius, U. 2004. The phylogenetic position of the comb jellies (Ctenophora) and the importance of taxonomic sampling. Cladistics 20: 558–578.
Wang, D. Y., Kumar, S. & Hedges, S. B. 1999. Divergence time estimates for the early history of animal phyla and the origin of plants, animals and fungi. Proceedings of Royal Society of London B 266: 163–171.
Wang, X. & Lavrov, D. V. 2007. Mitochondrial Genome of the Homoscleromorph Oscarella carmela (Porifera, Demospongiae) Reveals Unexpected Complexity in the Common Ancestor of Sponges and Other Animals. Molecular Biology and Evolution 24: 363–373. [pdf]
Wolf, Y. I., Rogozin, I. B., & Koonin, E. V. 2004. Coelomata and Not Ecdysozoa: Evidence From Genome-Wide Phylogenetic Analysis. Genome Research 14: 29–36. [pdf]